Com'era questo contenuto?
CelerisTx: scoperta di farmaci per malattie incurabili con ML su AWS
Sfruttare la scoperta di farmaci computazionale sui composti che inducono la prossimità (PIC)
L'umanità ha sviluppato nel tempo innumerevoli soluzioni terapeutiche per curare le malattie. Tecnologie recenti come il knockout (CRISPR — funziona a livello di DNA) e il knockdown (RNAi — funziona a livello di RNA) presentano limitazioni significative. Al contrario, i composti che inducono la prossimità (PIC) combinano i vantaggi unici delle piccole molecole. Tali caratteristiche benefiche includono la biodisponibilità orale e la facilità di produzione, insieme a quelle delle tecnologie di silenziamento come CRISPR e RNAi. A differenza di RNAi e CRISPR, i PIC influenzano le proteine, fornendo così una modalità terapeutica rivoluzionaria per colpire le proteine, specificamente associate alle malattie. Le alternative che prendono di mira le proteine includono farmaci biologici e inibitori, ma queste alternative non hanno ampliato la gamma di proteine patogene che possono essere prese di mira. Oltre l'80% delle proteine patogene è associato a una malattia non ancora suscettibile di interventi farmaceutici.
La degradazione proteica mirata (TPD) è la modalità farmacologica di induzione della prossimità più popolare, dimostrata per la prima volta all'inizio degli anni 2000. Ha anche ricevuto il premio Nobel per la chimica per la scoperta della degradazione delle proteine mediata dall'ubiquitina nel 2004. Il concetto di TPD implica la degradazione selettiva delle proteine dirottando i macchinari cellulari interni al corpo umano. Rispetto ai metodi basati sull'occupazione, come gli inibitori, i meccanismi TPD hanno il vantaggio di eliminare le funzioni dell'impalcatura e quindi di affrontare la causa principale del problema piuttosto che limitarsi a curare i sintomi. In generale, sono necessari strumenti per eliminare le proteine mal ripiegate e denaturate all'interno di una cellula per mantenere l'omeostasi biologica.
Sebbene tramite i PIC si possano utilizzare attivamente diversi macchinari, come i lisosomi e l'autofagia, Celeris Therapeutics si concentra sul sistema ubiquitina-proteasoma. In base a questo sistema, le proteine indesiderate vengono marcate con l'ubiquitina, una piccola proteina di segnalazione. Di conseguenza, questo processo avvia la degradazione delle proteine patogene.
Tuttavia, la valutazione del TPD in un laboratorio sperimentale è lenta e costosa. Celeris Therapeutics, pertanto, ha implementato un flusso di lavoro computazionale per prevedere efficacemente la degradazione delle proteine e quindi abbreviare e semplificare i tempi di sviluppo dei farmaci.
Composti che inducono la prossimità
Celeris Therapeutics (CelerisTx) è un pioniere nell'adozione dell'IA sui PIC, concentrandosi sulla degradazione proteica mirata. In termini di dati, nella degradazione delle proteine, ciò significa identificare più molecole che interagiscono tra loro in un modo particolare. Immagina i mattoncini LEGO®. Invece di unirli in base a una sola dimensione, ad esempio la forma, questi elementi hanno più dimensioni o requisiti da soddisfare. Alcuni sono di natura chimica, biologica, fisica, ecc., il che significa che le interazioni sono soggette a leggi specifiche. Queste leggi devono essere quantificate e incorporate nei dati. Esempi concreti di tali informazioni includono strutture 3D di molecole rappresentate come grafici, idrofobicità o potenziale elettrostatico. Complessivamente, ciò equivale a circa 20 diverse funzionalità curate dagli esperti TPD. Di conseguenza, viene generata un'enorme quantità di dati che devono essere analizzati utilizzando degli algoritmi di machine learning e, in particolare, delle pipeline geometriche di deep learning. È necessaria un'applicazione di ML concreta per determinare il grado di interazione tra le molecole coinvolte in un cosiddetto complesso ternario. In altre parole, considerando due proteine, ci aspettiamo un valore scalare che determini il grado di interazione. Oltre ad essere accurati e generalizzabili, dobbiamo fare previsioni rapide in un sistema performante. Mentre simuliamo diverse interazioni proteina-proteina, è necessario scartare rapidamente le previsioni imprecise per analizzare tempestivamente lo spazio. Questa si è rivelata una sfida per un set di dati di interazione con oltre 20.000 coppie di proteine.
In che modo AWS è stato sfruttato per vari progetti di scoperta PIC
Per far risparmiare tempo agli sviluppatori e ottenere informazioni più rapide sui problemi specifici relativi allo sviluppo del machine learning, ci siamo rivolti ad Amazon SageMaker. Le sue capacità ci hanno permesso di evitare di implementare noi stessi alcune soluzioni di infrastruttura di machine learning, come il rilevamento delle distorsioni o l'ottimizzazione degli iperparametri. Il rilevamento delle distorsioni era importante durante la preparazione dei dati; quindi, abbiamo sfruttato SageMaker Clarify. Questo è stato fondamentale per garantire la qualità dei nostri dati, prima ancora che iniziassimo a modellare. Successivamente, una volta iniziata la modellazione, è stato importante disporre di integrazioni dirette di librerie geometriche di deep learning, come SageMaker Deep Graph Library (DGL). Poiché DGL è un pacchetto python open source per il deep learning su grafici, lo abbiamo utilizzato per ottenere una configurazione rapida dell'infrastruttura necessaria per il deep learning geometrico, che è essenziale.
Una volta impostati i modelli iniziali, per trovare rapidamente i migliori iperparametri, abbiamo utilizzato SageMaker Automatic Model Tuning. Stimiamo di aver così evitato mesi di tempo di sviluppo nella codifica dei framework di ottimizzazione degli iperparametri. Abbiamo sfruttato SageMaker Experiments per tracciare e organizzare tutte le modifiche agli esperimenti. Questa soluzione è stata particolarmente importante. Consideriamo il già citato problema di determinare la forza dell'interazione tra le proteine coinvolte nel complesso ternario, le oltre 15 architetture geometriche di deep learning che abbiamo sperimentato a partire dai livelli di attenzione, gli operatori di pura convoluzione sui grafici, ecc. La tracciabilità dei diversi modelli e dei rispettivi parametri era importante per determinare cosa avrebbe funzionato alla fine. Una volta creata la versione finale del modello, dovevamo eseguirne il debug e ottimizzare ulteriormente la pipeline. A questo scopo, si è rivelato utile SageMaker Debugger.
Oltre alle soluzioni software di SageMaker, avevamo bisogno di una grande potenza di calcolo per le nostre vaste pipeline di dati e deep learning. Pertanto, abbiamo creato una serie di istanze spot ottimizzata per le GPU utilizzate nell'addestramento dei nostri flussi di lavoro di machine learning.
Ottimizzazione del costo della pipeline di ML
Le istanze spot sono un tipo speciale di istanza Amazon EC2 che offre uno sconto del 90% rispetto ai prezzi on demand. A volte, a causa dell'elevata richiesta di risorse informatiche, queste stesse risorse possono essere interrotte, causando la perdita di un'intera sessione di calcolo da parte dell'utente. È fondamentale salvare i risultati intermedi del calcolo, se si addestra un modello di deep learning per un lungo periodo o se sull'istanza spot è stato eseguito un calcolo necessariamente di lunga durata. Oltre a salvare i risultati intermedi del calcolo, l'utente dovrebbe anche essere in grado di continuare automaticamente il calcolo su un'altra istanza spot a partire dallo stesso passaggio intermedio in cui è avvenuta la disconnessione.
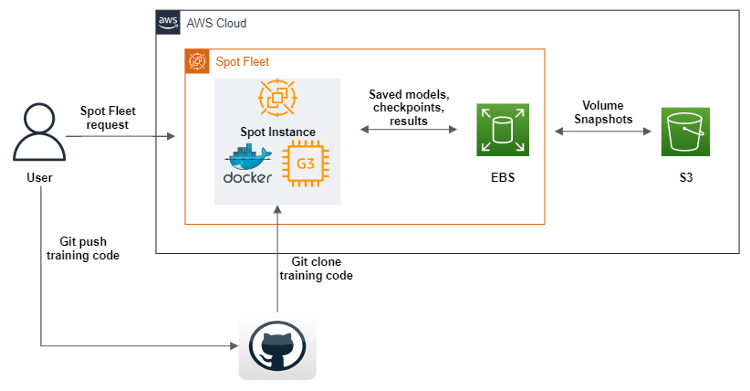
AWS SDK per Python (Boto3) è stato utilizzato per creare uno script che, dopo l'esecuzione, invierà una richiesta serie di istanze spot per creare un'istanza spot GPU con Amazon Machine Image (AMI), contenente tutto il software e le dipendenze per eseguire il nostro codice di machine learning. Inoltre, definiamo script di dati utente che scaricano un'immagine Docker da Amazon Elastic Container Registery (Amazon ECR) e quindi Git clona il codice dal nostro repository. Infine, gli script dei dati utente eseguono il codice geometrico della pipeline di deep learning. Questo codice è stato modificato in modo tale che, dopo ogni epoca di formazione sul machine learning, invii set di dati, log, modelli e checkpoint a un volume Amazon EBS. Per garantire che gli oggetti vengano salvati, li trasferiamo tutti su Amazon S3. Oltre a definire una serie di istanze spot, abbiamo anche abilitato il rifornimento di tutte le istanze spot che si disconnettono durante il processo, il che ci consente di continuare l'addestramento in modo automatico dagli ultimi checkpoint trovati nei bucket S3.
SageMaker offre qualcosa di molto simile, chiamato Managed Spot Training. Tuttavia, non abbiamo sfruttato l'offerta SageMaker perché, se utilizzata come parte di SageMaker, le istanze EC2 sono più costose di quelle standard. Ciò ha senso poiché SageMaker fornisce molte offerte di ML, facilità d'uso, meno tempo di progettazione necessario, ecc. Per noi, l'architettura di questa soluzione è stato un investimento strategico di tempo, perché sapevamo che avremmo utilizzato GPU con capacità diverse per diversi anni.
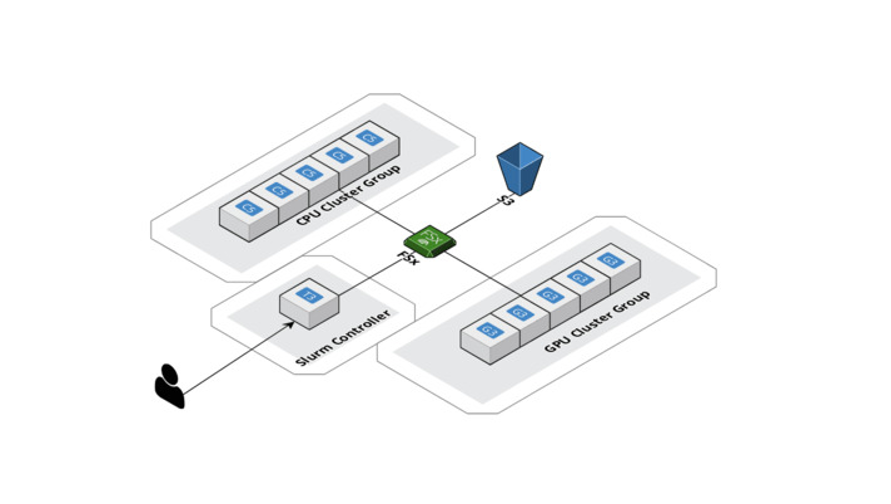
L'HPC era un altro caso d'uso significativo che dovevamo soddisfare con AWS. L'enorme spazio di dati che deve essere analizzato con il deep learning geometrico comporta un notevole onere computazionale. Abbiamo scalato i nostri calcoli orizzontalmente con AWS ParallelCluster e creato un sistema di file condiviso, Amazon FSx per Lustre, in modo che tutti i nodi di calcolo potessero accedere e modificare i dati dalla stessa posizione. Inoltre, AWS supporta Slurm come gestore del carico di lavoro, che ci consente di distribuire il lavoro tra CPU e GPU contemporaneamente. Ciò alla fine consente non solo una sperimentazione più rapida, ma anche una gestione ottimizzata dei modelli. Ancora una volta, va notato che SageMaker offre un servizio simile per set di dati e modelli di grandi dimensioni, chiamato Distributed Training Libraries.
Il futuro dei composti che inducono la prossimità
I risultati preliminari della nostra pipeline sono disponibili per la malattia di Parkinson che progredisce verso gli studi preclinici. Sperimentare in modo rapido e affidabile è essenziale per la scoperta di farmaci computazionale, poiché continuiamo a migliorare la nostra pipeline di ricerca sui farmaci con nuove architetture e approcci. Il nostro approccio a ricerca e sviluppo, che ci vede investire ingenti risorse nella ricerca, ci pone di fronte a molti rischi continui che dobbiamo mitigare. Le offerte standard di AWS sono un modo per garantire l'agilità e il passaggio più rapido dalla ricerca al laboratorio e poi al mercato.
Conclusioni
Ora è il momento di aumentare la produttività nella scoperta di farmaci. L'inverso della legge di Moore in farmacologia, nota come "Legge di Eroom", indica che i costi di ricerca e sviluppo necessari per sviluppare un nuovo farmaco continueranno ad aumentare. Un approccio per risolvere questo problema consiste nello sfruttare e semplificare il processo di esecuzione di esperimenti di machine learning sostenuti per la ricerca sui nuovi farmaci.

Christopher Trummer
Christopher Trummer è co-fondatore di Celeris Therapeutics e ricopre il ruolo di CEO. È stato più volte ospite come relatore principale in conferenze sull'IA applicata alla scoperta di nuovi farmaci e ha contribuito come coautore a pubblicazioni sottoposte a revisione paritaria su diverse riviste scientifiche.

Noah Weber
Noah Weber è Chief Technology Officer presso Celeris Therapeutics. È un Kaggle Grandmaster ed è anche un docente aggiunto presso l'Università di Tecnologia di Vienna e l'Università di Scienze Applicate di Vienna.

Olajide Enigbokan
Olajide Enigbokan è Startup Solutions Architect presso Amazon Web Services. Ama lavorare con le startup (soprattutto con i costruttori) per scoprire il valore del cloud AWS.

AWS Editorial Team
Il team Content Marketing di Startup AWS collabora con startup di varie dimensioni e in ogni settore, al fine di sviluppare contenuti eccezionali che siano informativi, coinvolgenti e autentici fonti di ispirazione.
Com'era questo contenuto?